2020 年 1 月,整個中國因新型冠狀病毒 2019-nCov 得了一場“感冒”。疫情一開始,武漢病毒研究所石正麗團隊就用實驗證實了血管緊張素轉化酶 2(Angiotensin-converting enzyme 2,ACE2)是新型冠狀病毒感染人體的受體基因。2020 年 1 月 26 日,上海同濟大學醫學院左為研究團隊在《bioRxiv》上發表了題為“Single-cell RNA expression profiling of ACE2, the putative receptor of Wuhan 2019-nCov”的文章。
該研究利用已有的數據庫,結合單細胞 RNA 測序技術中相關生信分析,對 ACE2 在人肺內單個細胞的表達情況進行了分析,共涉及 8 個樣本,43134 個細胞。結果表明:ACE2 受體主要在一部分(1% 左右)II 型肺泡上皮細胞(AT2) 中表達;同時發現這些 AT2 細胞除了表達病毒受體,還表達與病毒復制和傳播相關的基因,說明其很可能是冠狀病毒的靶細胞。可見,單細胞測序技術不僅是科研的利器,同時還為破解疫情做了應有的貢獻。那么,1 月份利用單細胞測序技術在基礎研究中還有哪些突破呢?
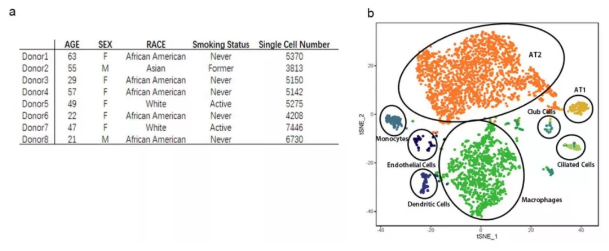
圖 1 人肺的單細胞分析
1 月份搜集到高分文獻(IF>10)26 篇,表 1 按照在線時間順序列出了文章所在期刊、研究方向,分選平臺以及英文題目等。
表 1 2020 年 1 月單細胞測序高分文獻集錦 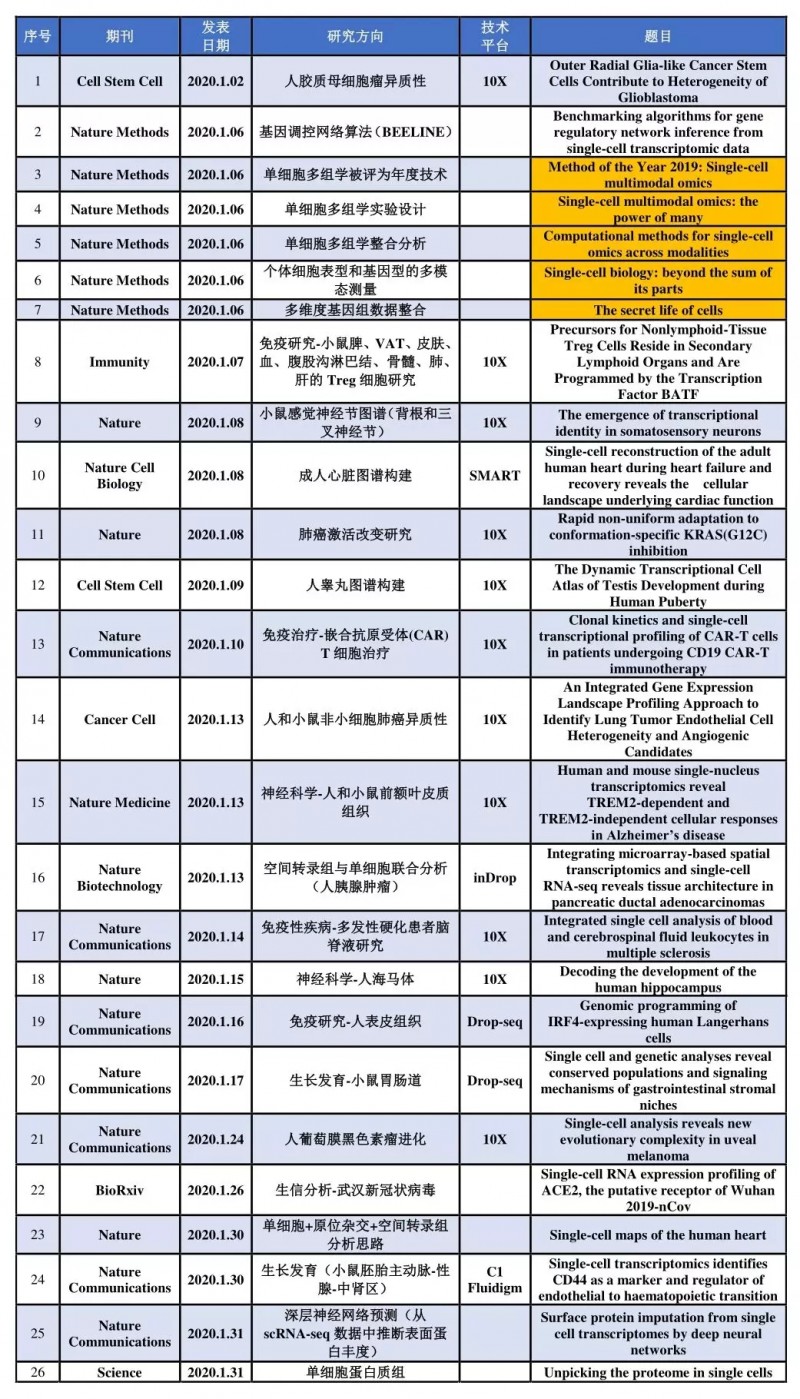
我們知道 2011 年,《Nature Methods》將單細胞測序列為當年度值得期待的技術之一;2013 年 1 月,《Science》雜志將單細胞測序列為年度值得關注的六大領域榜首;2014 年 1 月,《Nature Methods》將單細胞測序列為 2013 年度重要的方法學進展。讓人興奮的是 2020 年 1 月 6 號《Nature Methods》將單細胞多組學分析選為了“Method of the Year 2019”,并在線發表 5 篇相關文章(表 1 中標黃部分)。

其中,加利福尼亞大學圣地亞哥分校任兵教授在文章“Single-cell multimodal omics: the power of many”中分享了單細胞多組學的實驗設計。文章首先總結現有的單細胞多組學測序技術的應用范圍,包括細胞核內 DNA,RNA,DNA 甲基化,染色體開發程度,染色體三維結構,胞質 RNA,線粒體基因組,細胞表面蛋白質以及基于 CRISPR 的測序技術。
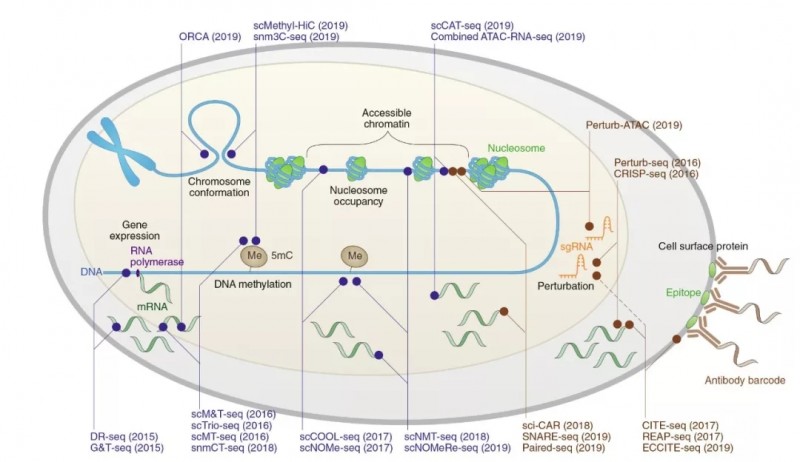
圖 2 已開發的同時分析單細胞表觀遺傳特征、DNA 序列、基因表達變異和細胞表面蛋白的方法。藍色代表低通量高成本的測序技術,棕色代表高通量低成本的測序技術。
然后作者總結現有技術的不足和發展的方向,比如單細胞基因檢測數較少(可通過實驗設計改善);單細胞蛋白組學不成熟(提高靈敏度和通量);空間測序技術只用于轉錄組(應擴展到其他組學)。
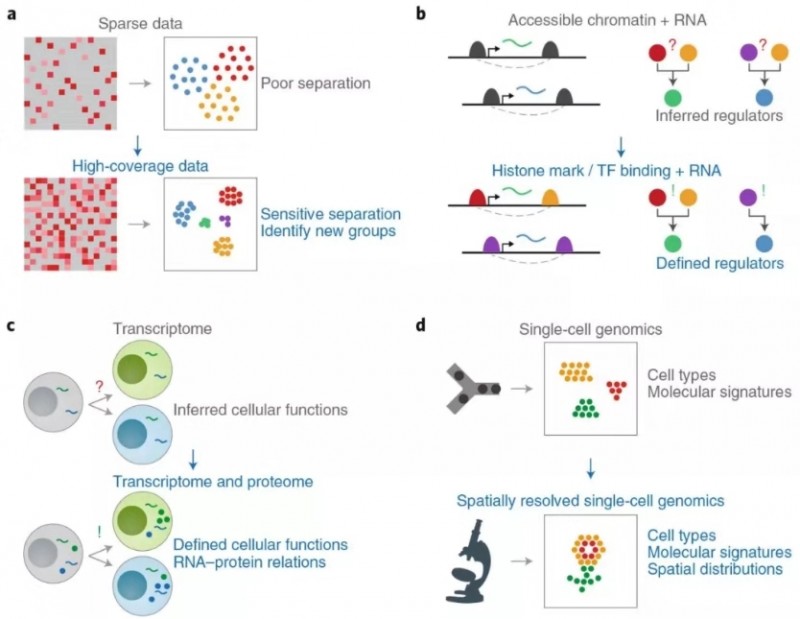
圖 3 現有單細胞多組學技術的不足和發展方向
同時在線的另外一篇文章“Computational methods for single-cell omics across modalities”則講述了單細胞多組學聯合分析方法。分析對象:整合不同數據對象(ATAC,RNA,蛋白)的同時,保留細胞類型固有生物學差異。實際問題:批次效應處理,ATAC/RNA 整合,CITE-seq 數據分析等。基本方法:CCA/ 非負矩陣分解 / 自編碼等。基本原理:多組學整合的思路與批次效應去除的思路相似,combat 是基于線性回歸的批次效應去除方法,CCA 是 Seurat 剛開始處理批次效應的基本方法,后來用于 ATAC/RNA 整合的方法 anchor 也是基于 CCA,ATAC/RNA 整合的方法也可以用非負矩陣分解。未來可期:RNA 和 ATAC 組合可以促進調控網絡研究;空間數據可以輔助細胞間相互作用的分析;整合 Bulk 數據和單細胞數據。

圖 4 單細胞多組學聯合分析不同計算方法的示意圖
1 月份單細胞測序另一個突破就是和空間轉錄組實現強強聯合,以獲得目標組織的三維空間轉錄組圖譜。2020 年 1 月 13 日,《Nature Biotechnology》上發表了“Integrating microarray-based spatial transcriptomics and single-cell RNA-seq reveals tissue architecture in pancreatic ductal adenocarcinomas”的文章。作者對 3 個胰腺導管癌(PDAC)組織 A、B、C 進行 scRNA-seq(indrop-seq),同時對 2 個主要樣本(A、B,各 3 張切片),4 個附加樣本(D、E、F 和 G,各 1 張切片),共 10 張切片做空間轉錄組(ST)。樣品 A 和 B 的 scRNA-seq 和 ST 的數據集信息通過多模式相交分析(Multimodal intersection analysis,MIA)進行整合,就是用超幾何算法檢驗細胞亞群的 marker 基因與組織亞區的 marker 基因重疊的數目是否達到顯著水平,若顯著,則認為該細胞亞群與該組織亞區相對應。
作者確定了組織中亞區中特定的細胞類型后,通過進一步細分細胞亞群和組織亞區,再次利用 MIA 算法進行映射,獲得了更為詳細的細胞位置 - 類型信息。作者利用得到的 MIA 結果繪制了不同腫瘤樣本微環境的特點,免疫環境狀態,應激水平以及細胞之間相互作用的模式,有助于預判患者預后。可見,MIA 算法的開發可將高分辨率的 scRNA-seq(單細胞水平)和帶空間位置信息的 ST 結合,為全面解析組織生物學信息提供了技術支撐。
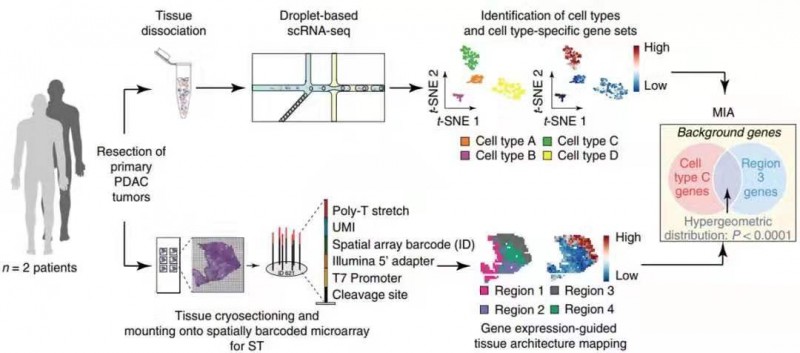
圖 5 實驗設計與分析示意圖。手術切除的 PDAC 腫瘤同時進行 scRNA-seq 和 ST 處理。聚類后,根據特異表達的基因推斷每個簇的細胞類型。剩余組織的冷凍切片用于 ST 分析,其中每個點捕獲組織中特定位置細胞中的轉錄組。將多模式相交分析(MIA)應用于兩個數據集,揭示了各種細胞類型的空間分布。

