2 月 11 日,世界衛生組織(WHO)正式將新冠肺炎病命名為 COVID-19,國際病毒分類委員會(ICTV)將新冠病毒命名為 SARS-CoV-2。SARS-CoV 病毒引起 SARS 病,SARS-CoV- 2 病毒引起 COVID-19 病,從官名上能看出新冠病毒與非典病毒有著密切聯系,科研人員也揭示了不同病毒間蛋白質(氨基酸序列)的差異可能是新冠病毒比 SARS 和 MERS 更具傳染性的原因。除了蛋白方面的差異,病毒基因組也必然存在著差異。從 2003 年的 SARS 到 2012 的 MERS,再到 2020 的 COVID-19,毫無疑問,我們肉眼看不到的冠狀病毒也在發生著變異與進化,這些變異有利于病毒自身,卻危害人類生命健康。今天小編要分享的便由變異所起——拷貝數變異的相關信息,提供拷貝數變異檢測方面的參考。
拷貝數變異的形成和致病機制
拷貝數變異屬于結構變異的一種,可分為拷貝數增加和拷貝數減少。拷貝數變異是人類遺傳多樣性的來源之一。而且通過重組、復制或其他方式產生的拷貝數變異似乎比單核苷酸多態性要高得多。但拷貝數變異并不一定會導致疾病,可能僅僅是作為一種多態性而存在,這與大量的單核苷酸多態性一樣,是良性的。異常的拷貝數變異通常是癌癥、遺傳病甚至是某些復雜疾病的重要分子機制。
拷貝數變異的產生主要涉及 4 種機制:
非同源重組、非同源連接、L1 反轉錄以及復制叉滯后連接等。
拷貝數變異對表型的影響機制主要有:
1)基因劑量;
2)功能失活;
3)基因融合;
4)位置效應;
5)橫向效應等。
拷貝數變異與三體綜合征
人類很早就認識到拷貝數變異與疾病之間的關系,但受到檢測精度的限制,早期的發現以大的染色體水平的變異為主。隨著基因芯片、高通量測序等多種檢測技術的快速發展,越來越多的拷貝數變異被檢測出來,包括亞顯微結構的拷貝數變異檢測成為可能。
三體綜合征是典型的基因組拷貝數變異引起的疾病,例如 21 三體綜合征、18 三體綜合征和 13 三體綜合征。21 三體綜合征又名唐氏綜合征,發病率約為 1 /700。13 三體綜合征于 1657 年被 Thomas Bartholin 初次記載,并由 Klaus Patau 在 1960 年報道染色體核型,也稱為 Patau 綜合征。18 三體綜合征于 1960 年被 John Hilton Edwards 報道,也被稱為 Edwards 綜合征。
拷貝數變異數據庫
拷貝數變異數據收錄于 DGV 數據庫(http://dgv.tcag.ca/dgv/app/home),記錄了一系列基因變異與表型相關的信息,數據庫信息持續更新,截至 2020.2.19,DGV 數據庫收錄了總共 72 項研究 6359956 個樣本的 CNV 信息,大多數的 CNV 分布在 1 -10Kb,其次為 100-500bp。
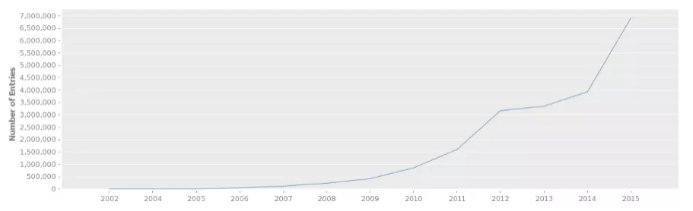
圖 1 DGV 數據庫變異數目逐年增加情況
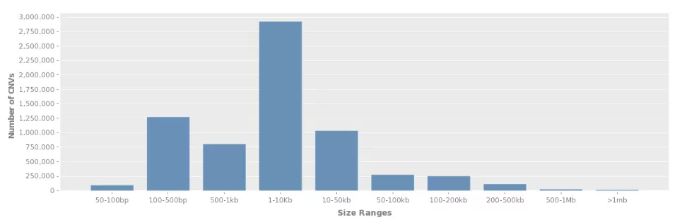
圖 2 DGV 數據庫 CNV 大小的分布情況
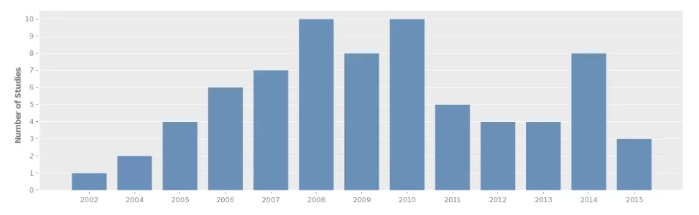
圖 3 DGV 數據庫項目數情況
拷貝數變異檢測平臺的技術比較
傳統的染色體核型分析、全基因組范圍內 CNV 芯片、高通量測序技術均可以對 CNV 進行檢測,接下來的兩篇文獻均是關于芯片和測序兩大 CNV 檢測平臺的比較研究,孰優孰劣讓我們一探究竟。
文獻 1 是 2016 年香港中文大學醫學院團隊發表于國際醫學遺傳學權威學術雜志 Genetics in Medicine 的文章討論了 NGS 技術是否可以作為常規臨床應用中 CNV 檢測的替代方法。

該團隊使用低覆蓋度的全基因組測序流程,對 570 名患者的多中心組進行了全基因組 CNV 分析(> 50 kb)和芯片檢測分析。根據美國醫學遺傳學和基因組學指南對 CNV(即致病性 CNV,pCNV)進行了分類。結果發現,CMA 和基于 NGS 的方法在 71 個驗證樣本中的 pCNV 的結果一致(如下圖表)。
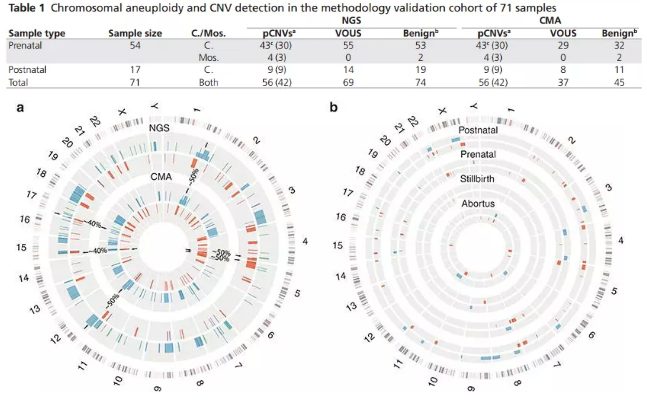
研究者也指出,高深度全基因組測序(whole genome sequencing,WGS)可以提供整個基因組的全面視圖,是一種高通量的檢測單核苷酸變異、插入 / 缺失、拷貝數變異和基因組結構重組的技術手段。但目前而言,高深度 WGS 的價格高昂,且變異信息較多,較難在產前診斷中廣泛應用;而基于低深度 WGS 的水平,能夠在降低每個樣本的測序成本基礎上,同時提高檢測通量,達到低深度高通量的目的(low-pass WGS)。
文獻 2 也是來自于同一團隊,2019 年 12 月份發表于 Genetics in Medicine,增加了樣本例數,對低深度高通量全基因組測序和染色體微陣列分析進行了深入比較,分析結果支持了將低深度全基因組測序用于產前診斷中以提供更多有意義的臨床信息。

研究人員將 2016-2019 年入組的 1023 名孕婦同時進行了 CMA 和 NGS 檢測。CMA 檢測出共 121 例樣本的異常,包括 87 個非整倍性和 37 個致病或可能致病的 CNV,這兩種染色體異常存在部分重疊;而低深度測序不僅檢測到了全部的 CMA 檢出的染色體異常,還額外檢測到 17 例非整倍性或致病 / 可能致病的染色體拷貝數變異。作者進一步分析了測序分析結果的靈敏度和特異性,以芯片分析為基準,低深度全基因組測序達到了 99.9%(121/121)的靈敏度和 87.7%(121/138)的特異性,總體診斷陽性率為 13.5%(138/1023)。在 DNA 起始量方面,芯片起始量 300ng DNA 和低深度測序起始量 50ng 為標準進行了樣本制備,結果是低深度全基因組測序樣本中有 5 例未能通過初期試驗,而芯片則有 47 例未通過,也就是造成了重復實驗率分別為 0.5%(5/1023)和 4.6%(47/1023),這些結果綜合說明低深度全基因組測序應用于產前診斷的樣本要求更低,實驗也更加穩定。
從 CNV 檢測技術總體來看,傳統的染色體核型分析一直被認為是確診染色體變異的標準,也是染色體病產前診斷的一線方法,但是檢測周期長、分辨率較低,無法檢出 5Mb 以下的 CNV。目前主要用于全基因組范圍內 CNV 檢測的“金標準”技術為 CNV 芯片,檢測范圍廣、分辨率較高;缺點是成本較高,在大規模的產前診斷應用中有所限制。而近年來發展起來的高通量測序技術,為 CNV 檢測提供了新的手段,具有成本低、通量高、檢測便捷等優勢。
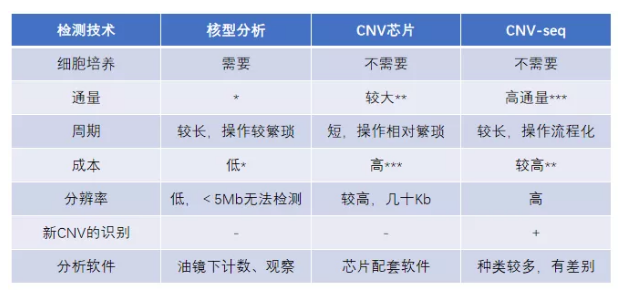
不可否認,芯片方法依然是拷貝數檢測的金標準,基于高通量測序平臺檢測 CNV 的多篇文獻中也提到了這一點,并以此為基準分析測序方法的準確度和靈敏度。高通量的方法作為后起之秀,檢測結果依賴于測序質量、測序數據量、分析軟件等,完全替代芯片還需些時間。伯豪生物提供芯片和高通量測量測序兩種平臺的檢測服務,小編建議各位老師們可以按需選擇。
這次疫情已導致 7 萬余人受到新冠病毒的感染,同時得到了國內外千千萬萬人的高度關注,我們有理由相信,人類終將贏得這場戰“疫”,而勝利之后的人類該會敬畏自然,善待自然界的其他動物們!


